热线:021-66110810,66110819
手机:13564362870


热线:021-66110810,66110819
手机:13564362870
细菌生物膜或称细菌生物被膜是指附着于有生命或无生命物体表面被细菌胞外大分子包裹的有一定三维结构和功能的细菌群体,是大多数微生物在自然界中采用的一种生活方式[1]。生物膜的结构复杂,内部分布不均匀。生物膜的结构与细菌种属和环境条件息息相关,不同细菌或同一细菌所处环境不同时,生物膜的疏密和厚薄都会存在明显差异。由于生物膜中的细菌处在不同时间和空间发展,因此基因表达和生理活性具有不均质性。此外,生物膜具有较高的耐药性[2]、抗吞噬性[3]以及极强的黏附性[4],可以导致多种细菌感染性疾病,是细菌感染的重要根源之一。与生物膜相关的细菌感染性疾病的治疗极为棘手,目前主要有2种策略:一是抑制生物膜的形成,如应用表面镀银和含钛导管;二是用抗菌药物来治疗已形成的生物膜,如应用抗菌肽(antimicrobial peptides,AMPs)、AMPs与抗生素联用或借助纳米递药系统。
1生物膜的形成
生物膜的形成是一个复杂的动态过程,主要可分为5个阶段:1)可逆附着阶段,细菌非特异性地附着在底物表面上;2)不可逆附着阶段,细菌与底物表面通过黏附素蛋白或黏附因子相互作用;3)微菌落形成阶段,细菌产生胞外聚合物;4)生物膜成熟阶段,细菌合成并释放信号分子;5)细菌脱落/扩散阶段,细菌离开生物膜,回归独立的浮游生活方式。以猪链球菌生物膜形成过程为例,如图1所示。
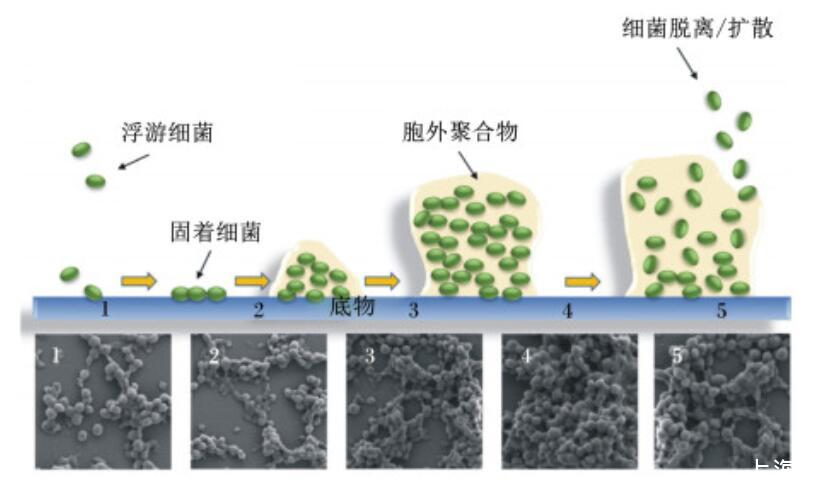
1:可逆附着reversible attachment;2:不可逆附着irreversible attachment;3:微菌落的形成micro colony;4:成熟maturation;5:细菌脱离/扩散cellular detachment。图1猪链球菌生物膜形成过程示意图和扫描电镜图
1.1生物膜的黏附
细菌在刚接触物体表面时会借助布朗运动、重力、扩散、对流或内在运动来进行可逆性的黏附。当细菌与基质表面之间产生的吸引力大于排斥力时,细菌就会在基质表面完成附着,但当吸引力小于排斥力时,细胞则无法实现附着,从表面脱落,恢复浮游状态。关于细菌如何实现可逆附着,目前存在2种不同的主张:一是认为细菌需要借助黏附素蛋白来完成初始的可逆附着,这一观点中强调了黏附素蛋白的特异性选择,其受体为宿主表面的蛋白、糖蛋白和糖脂。如多糖细胞间黏附素(polysaccharide intercellular adhesion,PIA)是表皮葡萄球菌在医疗器械上定植的关键物质[6];二是认为细菌的不可逆附着过程中不存在特异性选择,其依靠的是细菌表面的附属结构及黏附因子的黏合作用,细菌的附属结构常具有较高的亲和性,有试验证明Ⅳ型纤毛等都具有黏性末端,可以帮助细菌完成黏附[7],而鞭毛可以加快细菌和表面进行接触[8]和克服细菌与表面的排斥力。
细菌的可逆性附着会随着时间的推移变为不可逆附着,不可逆附着是通过短程相互作用实现的,例如偶极-偶极相互作用、氢键、离子键和共价键。不可逆附着可避免细菌本身被带到不利于自身生长的环境中,因此,生物膜一旦形成便很难消除。
1.2微菌落的形成
细菌黏附到表面后便开始生长繁殖,同时基因表达上的调整会促使其分泌大量的胞外聚合物(extracellular polymeric substance,EPS),其主要由多糖、蛋白质和细胞外脱氧核糖核酸(eDNA)组成。EPS构成了生物膜三维结构的框架,其可以黏附单个细菌并形成扁平或类似“蘑菇状”的微菌落,大量菌落堆积使生物膜加厚,进一步增强了细菌对环境(如抗菌剂、免疫因子、温度和竞争微生物)的适应能力[9]。因此,EPS分子的产生对生物膜结构十分重要。生物膜的发展也离不开细菌内的信号系统,有研究证明,环二鸟苷酸(c-di-GMP)是细菌在浮游状态和固着状态之间相互转变的关键信号分子之一,可以调控多种细菌生物膜内EPS的分泌和转运[10]。高浓度的c-di-GMP会正向调节生物膜的形成,而低浓度的c-di-GMP会对生物膜的形成产生负向影响[11]。
1.3生物膜的成熟
成熟的生物膜微菌落之间围绕着输水通道,可以运送养料、酶、代谢产物和排出废物等[12-13]。这一过程中常伴随着群体感应(quorum sensing,QS)调控系统,当细菌合成的信号分子浓度达到阈值水平后,位于细胞质或细胞膜中的受体会进行信号分子的识别,并激活相关的基因表达。如群体信号分子AI-2不仅可以促进不可分型流感嗜血杆菌生物膜增厚和成熟,而且在生物膜的发育和成熟过程中起到防止或延迟生物膜扩散的作用[14]。研究表明,铜绿假单胞菌通过释放群体感应分子(3-氧十二烷酰高丝氨酸内酯)抑制宿主免疫,从而更好的存活,并且3-氧十二烷酰高丝氨酸内酯在生物膜中细菌内积累浓度远高于浮游细菌内的浓度[15-16],这更直接地表明了群体感应在铜绿假单胞菌生物膜形成中的作用。
1.4细菌脱落/扩散
细菌在浮游状态和附着于多细胞群落的生物膜状态之间交替存在[17]。当生物膜成熟后,细菌从小菌落中分离出来,移动到新的底物上。这个过程对于生物膜来说也是一种很有效的传播方式,释放出的浮游细菌可以进一步的繁殖、迁移,从而形成新的生物膜。扩散是延续生物膜的一种策略,即生物膜内的细菌为适应特定的生理或环境的变化而启动的脱落行为[18-19]。
2生物膜的耐药机制
当浮游细菌形成生物膜后,细菌耐药性会大幅度增加,Shenkutie等[20]研究了黏菌素、环丙沙星和亚胺培南对9株鲍曼不动杆菌的最小杀菌浓度(MBC)和最小生物膜清除浓度(MBEC),结果表明9个菌株的生物膜细菌产生了高水平的抗生素耐药性,与浮游细菌相比,根除鲍曼不动杆菌生物膜需要将黏菌素浓度提高64倍,环丙沙星和亚胺培南浓度提高1 024倍。因此,若想预防或控制生物膜形成,耐药性是不容忽视的难题。关于生物膜的耐药机制主要存在以下几种假说。
2.1抗菌药物渗透障碍
生物膜对抗菌药物产生耐药性的主要原因之一是药物渗透性差,这是由于大多数抗菌药物扩散和吸附到EPS的能力减少[21](图2)。
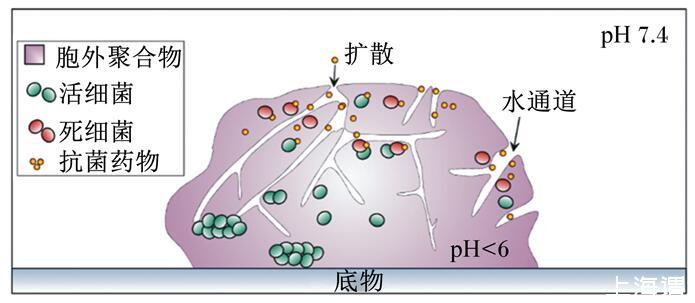
图2生物膜中的抗菌药物的渗透障碍
大多数抗菌药物受到大小限制难以通过水通道,因此对于包埋于EPS中的细菌来说,EPS相当于一个可以阻碍抗菌药物渗透的保护屏障,这种阻碍作用会降低与细菌接触的抗菌药物的浓度,从而使药物达不到有效浓度,反而造成了细菌耐药性的提高。但这一假说并不适用于所有生物膜,如抗生素妥布霉素和环丙沙星可以穿透生物膜,但未能实现有效杀死细菌[22]。此外,由于细菌及其分泌的黏性基质均带负电,所以亲水性抗菌药物会吸附在表面并形成电荷屏障,阻碍药物进入菌体发挥作用[23]。
2.2营养物质限制
细菌所处微环境的pH和氧化还原电位等不同,可使遗传学上一致的细菌个体表现不同的特性,如产生毒素不同可使生物膜中一些细菌个体对宿主无危害,而另一些细菌则可能对宿主有致命威胁。因此,不均质性是细菌生物膜的另一个重要特性,也与细菌的抗性有关。有研究表明,生物膜中的营养物质、氧和代谢产物浓度呈现出一种自外向内的下降趋势[24]。这种特殊的微环境和营养条件导致位于生物膜顶部的细胞具有较好的代谢活性,而其他细菌则处于稳定期或生长缓慢[12](图3),从而降低了对抗菌药物的敏感性。部分抗菌药物可以杀灭最外层敏感性较高的细菌,但只能部分杀灭或无法杀灭敏感性低的细菌。而死亡细菌会成为残存细菌的营养来源,从而形成新的生物膜。
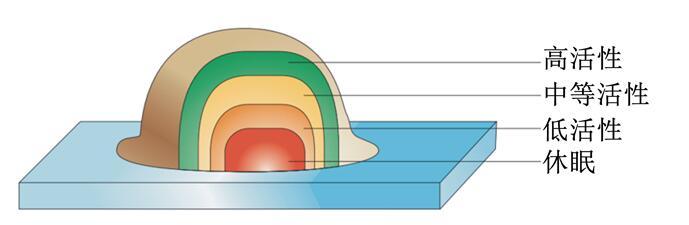
图3生物膜中细菌的代谢活性
2.3生物膜中基因表型改变
在形成生物膜时,细菌会由浮游状态转变成固着状态,这种生长环境的变化会导致浮游细菌和生物膜细菌之间存在表型差异,具体表现为这2种生活方式的细菌不共享相同的转录组或蛋白质组,生物膜内细菌对抗菌药物的敏感性远低于基因相同的浮游细菌。且生物膜中的细菌比浮游细菌积累突变的速度更快,这些突变会导致抗生素耐药性的增加[25]。生物膜中积累的突变会加速耐药菌株的出现,开发根除生物膜药物刻不容缓。
2.4产生QS信号
生物膜中的细菌可以利用QS机制产生耐药性[26]。当生物膜内的细菌数量超过一定阈值时,QS会使一部分细菌脱离表面,转为浮游态,导致感染扩散或复发。例如,低剂量的外源AI-2可显著增强猪链球菌的生物膜形成能力,促进耐药性的产生[27]。目前,有研究证明“淬灭”生物膜中的QS系统可增加生物膜对抗菌药物的敏感性,如群体感应抑制剂可以降低铜绿假单胞菌对妥布霉素的抗性,但不影响浮游细菌的敏感性[28]。所以,通过抑制QS系统也可以有效控制细菌生物膜形成。
2.5激活严紧反应
严紧反应是细菌的一种自我保护机制,可以确保细菌在恶劣的外界环境下存活。当缺乏合成蛋白质所必需的氨基酸时,细菌便会停止合成核糖体RNA,发生空转反应,RelA和SpoT调控警报素四磷酸鸟苷(ppGpp)和五磷酸鸟苷(pppGpp)的合成,从而引发细菌的适应性调控[29]。严紧反应在介导生物膜耐药性上的重要性已经得到证实,如氨基酸和葡萄糖缺乏会导致大肠杆菌对氧氟沙星和氨苄青霉素的长期耐受性[30]。Honsa等[31]分离出1株由于RelA错义突变导致ppGpp水平升高的耐万古霉素屎肠球菌(VRE)菌株,当在生物膜中生长时,它显示出对2种抗生素(托霉素和利奈唑胺)的极高的耐药性。因此,这种依赖于对饥饿条件的生理适应会影响生物膜内细菌对抗菌药物的敏感性,是生物膜产生耐药性的重要因素之一。
2.6启动外排泵系统
研究显示,生物膜内细菌中编码外排泵基因的相对表达量和浮游细菌中的表达量之间存在差异。氟康唑在白色念珠菌生物膜形成早期诱导外排泵编码基因过度表达[32];在鲍氏不动杆菌生物膜形成的不同阶段,编码外排泵基因的表达均上调[33]。由此可以说明,外排泵系统参与了生物膜的形成过程,即细菌通过外排泵系统将抗菌药物泵出胞外,造成菌体内药物浓度下降。因此,外排泵系统与细菌形成生物膜后的耐药性密切相关。
2.7分泌抗生素水解酶
分泌抗生素水解酶也是产生耐药性原因之一,如铜绿假单胞菌生物膜基质中分泌的β-内酰胺酶,可以降解抗菌药物,从而阻止这些药物发挥作用。Bowler等[34]认为,基质中β-内酰胺酶的数量增加会导致成熟的铜绿假单胞菌生物膜对头孢他啶和美罗培南更具耐药性。
3 AMPs抗生物膜作用3.1 AMPs
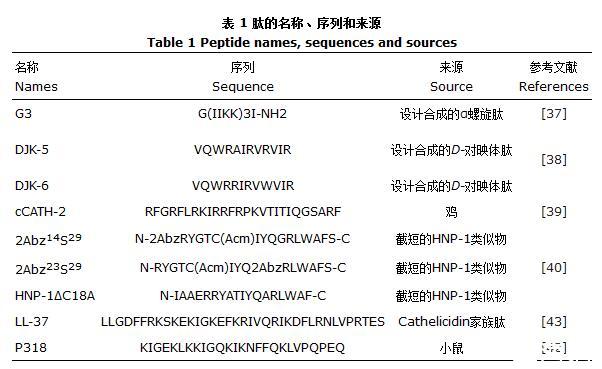
AMPs具有良好的抗生物膜活性,可以抑制和清除生物膜[35-36]。与传统抗生素相比,AMPs不易产生耐药性,是极具潜力的抗生素替代物。如螺旋肽G3可以干扰变形链球菌生物膜形成的不同阶段来抑制变异链球菌生物膜的形成。在初始阶段,G3通过降低细菌表面电荷、疏水性、膜完整性和黏附相关基因转录来抑制细菌黏附。在后期,G3与EPS中的eDNA相互作用,破坏成熟生物膜的3D稳定性结构,从而分散它们。因此,G3在预防和治疗变形链球菌生物膜感染方面表现出了巨大的应用潜力[37]。在铜绿假单胞菌生物膜开始生长时添加肽DJK-5和DJK-6,能够抑制生物膜的形成;在生物膜的结构已经明显时,DJK-5和DJK-6可以分散和根除成熟生物膜中的细菌[38]。肽cCATH-2对浮游菌和生物膜内细菌有快速杀灭能力,可以减少细菌最初附着和其他阶段的生物量[39]。Moazzezy等[40]发现,3种人中性粒细胞肽类似物均显示出对大肠杆菌生物膜的抑制和根除作用。抗菌肽LL-37在浓度远小于其MIC时即可破坏鲍曼不动杆菌生物膜,且随着LL-37浓度的增加,生物膜呈现出递减的趋势[41]。P318在0.15μmol/L时可以抑制白色念珠菌生物膜的形成[42]。上述肽的序列和来源详见表1。
表1肽的名称、序列和来源
3.2 AMPs与抗生素联用
部分AMPs与抗生素联用可以表现出很强的协同作用。青霉素、氨苄青霉素和红霉素与一系列含色氨酸的AMPs可以协同抑制表皮葡萄球菌生物膜的形成[44]。肽DJK-5和DJK-6与几种常见抗生素(环丙沙星、头孢他啶、亚胺培南和妥布霉素)联合使用时,抑制生物膜所需的抗生素浓度显著降低,其中DJK-5与环丙沙星联合抑制铜绿假单胞菌生物膜的FICI值为0.14(FICI指数为0.5表示良好的协同作用,相当于每个化合物组合使用时MBIC下降了4倍,FICI指数为1.0表示每个化合物组合使用时MBIC下降了2倍),DJK-6与头孢他啶联用抑制大肠杆菌0157的FICI值为0.35[38]。与生理盐水组相比,DJK-5和抗生素的联合使用明显减少小鼠皮肤脓肿模型中的细菌总数,表现出较好治疗效果[45]。Zhang等[46]研究了14种抗生素联合CRAMP对铜绿假单胞菌生物膜形成的干预作用,最终筛选出以1/4最低抑菌浓度的CRAMP与黏杆菌素结合可以显著抑制铜绿假单胞菌生物膜内的活菌数和生物量。Dosler等[47]研究发现,3种抗生素(妥布霉素、环丙沙星和黏菌素)分别与LL-37和CAMA存在协同作用,可以抑制铜绿假单胞菌生物膜的形成,其中环丙沙星与LL-37/CAMA的协同效果最好。肽AS10在0.22μmol/L时可以抑制白色念珠菌生物膜,并与两性霉素B和卡泊芬净协同作用于成熟生物膜,FICI值小于或等于0.5[42]。Chernysh等[48]发现,肽FLIP7与美罗培南、阿米卡星、卡那霉素、氨苄青霉素、万古霉素和头孢噻肟有高度协同作用,FICI值小于0.25,且与单独使用抗生素相比,用FLIP7和抗生素组合处理后的金黄色葡萄球菌和大肠杆菌生物膜内的细胞外基质、死细胞和活细胞都明显减少。莫匹罗星与蜂毒素的协同作用为消除金黄色葡萄球菌生物膜也提供了新的思路[49]。
鉴于生物膜的结构和特点,仅用抗生素很难将其彻底清除,因此将AMPs与传统抗生素联合使用具有完全消除生物膜的可能性,适合用于治疗生物膜导致的感染。但目前对于AMPs与抗生素联用的研究仍然停留在效应的观察上,缺乏联合作用机制的研究与抗体内生物膜研究。
3.3纳米递药系统
采用纳米递药系统可以克服AMPs在毒性、对蛋白水解的敏感性和药代动力方面的不足[50]。纳米粒根据材料来源可分为无机纳米粒载体和脂质纳米粒载体两大类(表2)。
表2纳米粒分类

近几年对无机纳米粒的研究相对较多,如银纳米粒本身就具有较强的抗菌活性和较强的抗菌膜效果,将其和AMPs结合后还可以提高AMPs的活性[51-52]。Gao等[53]研究开发了一种可以增强肽P-13抗菌活性的银纳米粒子,其可以提高肽P-13对4种细菌的抗菌活性[53]。脂质纳米载体可以改善药物的生物利用度和吸收稳定性,实现细菌膜靶向作用,是极有发展前途的新型药物传递系统,如脂质体是最常见脂质纳米载体。Meng等[54]通过在脂质体上修饰靶向基团实现了对EPS的选择性结合,进而提高了脂质体对细菌生物膜的靶向性结合。因此,纳米粒和AMPs联合治疗代表了开发抗生物膜药物的一种新方法。
4 AMPs抗生物膜机制4.1破坏或降解生物膜内细菌的细胞膜电位
革兰氏阴性菌EPS多由细胞外基质多糖组成,如脂多糖、蛋白聚糖、细胞外DNA。这些分子大部分带负电荷,并且通过电荷排斥和空间位阻抑制抗菌剂的扩散[55]。目前研究发现,大多数AMPs是通过引起细胞质膜去极化,诱导磷脂不规则分布,抑制重要代谢产物和细胞组分的合成,最终达到杀菌目的[56]。对3种不同结构的细菌素(乳酸链球菌素nisin A、乳链球菌素lacticin Q和羊毛硫细菌素nukacin ISK-1)的活性和作用方式进行研究,结果表明它们可以破坏膜电位,导致ATP外流,从而杀灭生物膜细菌[57]。
4.2生物膜基质的降解
AMPs还可以作用于生物膜的胞外聚合物基质,抑制其产生和积累。例如,肽PI可以降解变异链球菌产生的胞外多糖,导致聚苯乙烯或唾液包被的羟基磷灰石上形成的生物膜减少[58]。与上述结论类似,在人肝源性抗菌肽Hepcidin 20(Hep20)存在下,生物膜的结构发生了变化,细胞外基质明显减少[59]。
4.3避免细菌严紧反应
目前,已有研究表明,在(p)ppGpp缺乏的菌株中,细菌形成生物膜的能力会明显减弱。因此,通过调控(p)ppGpp合成酶和水解酶的表达来避免细菌严紧反应是抗生物膜的一个有效机制。如SpoT是一种双功能(p)ppGpp酶,即在营养物质缺乏的条件下SpoT会促进(p)ppGpp合成,在营养物充足时SpoT水解(p)ppGpp以下调严紧反应。肽1018通过下调SpoT的表达,导致(p)ppGpp降解,从而影响生物膜的形成[60]。
4.4干扰信号系统和下调负责生物膜形成的基因
抑制c-di-GMP的合成可以干扰生物膜的形成。Foletti等[61]研究发现,富含脯氨酸的短肽能够选择性地结合细菌第二信使c-di-GMP,从而可以抑制铜绿假单胞菌生物膜的生长。AMPs抑制生物膜形成的另一个机制是影响QS系统,例如LL-37降低了铜绿假单胞菌中与QS相关的基因表达,所以LL-37在低于抑制浓度时可以防止生物膜形成[62-63]。
在形成葡萄球菌生物膜时,PIA是表皮葡萄球菌生物膜发展过程中至关重要的物质,其可以使表皮葡萄球菌相互黏附,从而形成生物膜。PIA由icaABCD操纵子编码,而icaADBC操纵子由上游基因icaR编码的阻遏物负调控[64-65]。有研究发现,暴露于6倍MIC的hBD-3的菌株组显著下调了icaD的表达并上调了icaR的表达,而抑制icaD的转录会导致PIA的下调,从而减少了生物膜的形成[66]。
5小结
近年来,随着对细菌生物膜的研究,人们逐渐意识到生物膜感染会造成严重的危害。然而传统抗生素的治疗效果较差,且极易造成病原菌的耐药性,因此急需开发新型治疗生物膜感染的药物。AMPs具有多种积极的抗生物膜机制,可以抑制或消除生物膜,将其与抗生素联用可有效减少抗生素的用量,因此AMPs在治疗生物膜感染方面具有广阔的发展前景。然而,目前在实际临床治疗中仍存在以下问题亟待深入研究:AMPs自身对血清、蛋白酶敏感和生物安全问题,并且造成感染的常是已经成熟的生物膜。所以应重点解决AMPs自身存在的不足,并加强AMPs对已经成熟的生物膜的清除作用,以便于将AMPs更好的应用于相关治疗。纳米递药系统可以在一定程度上克服上述缺点,是开发AMPs抗生物膜药物未来研究的新方向之一。此外,继续深入研究AMPs如何影响生物膜信号传导、相关基因表达以及作用靶点等问题有助于更合理高效的预防、控制传染性和致病性生物膜形成,为开发治疗生物膜感染药物奠定理论基础。
参考文献
[1]
FLEMMING H C,WUERTZ S.Bacteria and archaea on Earth and their abundance in biofilms[J].Nature Reviews Microbiology,2019,17(4):247-260.DOI:10.1038/s41579-019-0158-9
[2]
CIOFU O,TOLKER-NIELSEN T.Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P.aeruginosa can escape antibiotics[J].Frontiers in Microbiology,2019,10:913.DOI:10.3389/fmicb.2019.00913
[3]
王贵年,范莹,王龙梓,等.黄芩苷对铜绿假单胞菌生物膜的影响[J].医学研究杂志,2011,40(1):131-132,143.
WANG G N,FAN Y,WANG L Z,et al.Effect of baicalin on Pseudomonas aeruginosa biofilms[J].Journal of Medical Research,2011,40(1):131-132,143(in Chinese).DOI:10.3969/j.issn.1673-548X.2011.01.039
[4]
ANNOUS B A,FRATAMICO P M,SMITH J L.Scientific status summary[J].Journal of Food Science,2009,74(1):R24-R37.DOI:10.1111/j.1750-3841.2008.01022.x
[5]
YI L,JIN M Y,LI JP,et al.Antibiotic resistance related to biofilm formation in Streptococcus suis[J].Applied Microbiology and Biotechnology,2020,104(20):8649-8660.DOI:10.1007/s00253-020-10873-9
[6]
MARINCOLA G,JASCHKOWITZ G,KIENINGER A K,et al.Plasmid-chromosome crosstalk in Staphylococcus aureus:a horizontally acquired transcription regulator controls polysaccharide intercellular adhesin-mediated biofilm formation[J].Frontiers in Cellular and Infection Microbiology,2021,11:660702.DOI:10.3389/fcimb.2021.660702
[7]
SERRA D O,RICHTER A M,KLAUCK G,et al.Microanatomy at cellular resolution and spatial order of physiological differentiation in a bacterial biofilm[J].MBIO,2013,4(2):e00103-e00113.
[8]
WOOD T K.Precedence for the structural role of flagella in biofilms[J].MBIO,2013,4(2):e00213-e00225.
[9]
LIMOLI D H,JONES C J,WOZNIAK D J.Bacterial extracellular polysaccharides in biofilm formation and function[J].Microbiology Spectrum,2015,3(3).
[10]
SCHÄPER S,STEINCHEN W,KROL E,et al.AraC-like transcriptional activator CuxR binds c-di-GMP by a PilZ-like mechanism to regulate extracellular polysaccharide production[J].Proceedings of the National Academy of Sciences of the United States of America,2017,114(24):E4822-E4831.DOI:10.1073/pnas.1702435114
[11]
KALIA D,MEREY G,NAKAYAMA S,et al.Nucleotide,c-di-GMP,c-di-AMP,cGMP,cAMP,(p)ppGpp signaling in bacteria and implications in pathogenesis[J].Chemical Society Reviews,2013,42(1):305-341.DOI:10.1039/C2CS35206K
[12]
DAVIES D.Understanding biofilm resistance to antibacterial agents[J].Nature Reviews Drug Discovery,2003,2(2):114-122.DOI:10.1038/nrd1008
[13]
JAMAL M,AHMAD W,ANDLEEB S,et al.Bacterial biofilm and associated infections[J].Journal of the Chinese Medical Association:JCMA,2018,81(1):7-11.DOI:10.1016/j.jcma.2017.07.012
[14]
PANG B,ARMBRUSTER C E,FOSTER G,et al.Autoinducer 2(AI-2)production by nontypeable Haemophilus influenzae 86-028NP promotes expression of a predicted glycosyltransferase that is a determinant of biofilm maturation,prevention of dispersal,and persistence in vivo[J].Infection and Immunity,2018,86(12):e00506-e00518.
[15]
SONG D K,MENG J C,CHENG J,et al.Pseudomonas aeruginosa quorum-sensing metabolite induces host immune cell death through cell surface lipid domain dissolution[J].Nature Microbiology,2019,4(1):97-111.DOI:10.1038/s41564-018-0290-8
[16]
TURKINA M V,VIKSTRÖM E.Bacteria-host crosstalk:sensing of the quorum in the context of Pseudomonas aeruginosa infections[J].Journal of Innate Immunity,2019,11(3):263-279.DOI:10.1159/000494069
[17]
BRIDGES A A,FEI C,BASSLER B L.Identification of signaling pathways,matrix-digestion enzymes,and motility components controlling Vibrio cholerae biofilm dispersal[J].Proceedings of the National Academy of Sciences of the United States of America,2020,117(51):32639-32647.DOI:10.1073/pnas.2021166117
[18]
ABDALLAH M,BENOLIEL C,DRIDER D,et al.Biofilm formation and persistence on abiotic surfaces in the context of food and medical environments[J].Archives of Microbiology,2014,196(7):453-472.DOI:10.1007/s00203-014-0983-1
[19]
LEE K,YOON S S.Pseudomonas aeruginosa biofilm,a programmed bacterial life for fitness[J].Journal of Microbiology and Biotechnology,2017,27(6):1053-1064.DOI:10.4014/jmb.1611.11056
[20]
SHENKUTIE A M,YAO M Z,SIU G K,et al.Biofilm-induced antibiotic resistance in clinical Acinetobacter baumannii isolates[J].Antibiotics(Basel,Switzerland),2020,9(11):817.
[21]
LIU Y,SHI L,SU L,et al.Nanotechnology-based antimicrobials and delivery systems for biofilm-infection control[J].Chemical Society Reviews,2019,48(2):428-446.DOI:10.1039/C7CS00807D
[22]
WALTERS M C,ROE F,BUGNICOURT A,et al.Contributions of antibiotic penetration,oxygen limitation,and low metabolic activity to tolerance of Pseudomonas aeruginosa biofilms to ciprofloxacin and tobramycin[J].Antimicrobial Agents and Chemotherapy,2003,47(1):317-323.DOI:10.1128/AAC.47.1.317-323.2003
[23]
魏建仝,钱军,苏秦柳晔,等.细菌生物膜引起致病菌耐药机制及抗菌肽LL-37对生物膜作用的研究进展[J].河西学院学报,2020,36(5):38-43.
WEI J T,QIAN J,SU Q L Y,et al.Mechanisms of resistance to pathogenic bacteria induced by bacterial biofilms and progress in the study of the effect of antimicrobial peptide LL-37 on biofilms[J].Journal of Hexi University,2020,36(5):38-43(in Chinese).
[24]
陈铁柱,李晓声,曾文魁,等.细菌生物膜耐药机制的研究与进展[J].中国组织工程研究与临床康复,2010,14(12):2205-2208.
CHEN T Z,LI X S,ZENG W K,et al.Research and progress of drug-resistant mechanism of bacterial biofilm[J].Journal of Clinical Rehabilitative Tissue Engineering Research,2010,14(12):2205-2208(in Chinese).
[25]
HALL C W,MAH T F.Molecular mechanisms of biofilm-based antibiotic resistance and tolerance in pathogenic bacteria[J].FEMS Microbiology Reviews,2017,41(3):276-301.DOI:10.1093/femsre/fux010
[26]
SAXENA P,JOSHI Y,RAWAT K,et al.Biofilms:architecture,resistance,quorum sensing and control mechanisms[J].Indian Journal of Microbiology,2019,59(1):3-12.DOI:10.1007/s12088-018-0757-6
[27]
YI L,LI J P,LIU B B,et al.Advances in research on signal molecules regulating biofilms[J].World Journal of Microbiology and Biotechnology,2019,35(8):130.DOI:10.1007/s11274-019-2706-x
[28]
BRACKMAN G,COS P,MAES L,et al.Quorum sensing inhibitors increase the susceptibility of bacterial biofilms to antibiotics in vitro and in vivo[J].Antimicrobial Agents and Chemotherapy,2011,55(6):2655-2661.DOI:10.1128/AAC.00045-11
[29]
张腾飞,罗青平,邵华斌,等.细菌的严谨反应研究进展[J].湖北畜牧兽医,2019,40(12):14-16.
ZHANG T F,LUO Q P,SHAO H B,et al.Advances in the rigorous reaction of bacteria[J].Hubei Journal of Animal and Veterinary Sciences,2019,40(12):14-16(in Chinese).DOI:10.3969/j.issn.1007-273X.2019.12.005
[30]
POOLE K.Stress responses as determinants of antimicrobial resistance in gram-negative bacteria[J].Trends in Microbiology,2012,20(5):227-234.DOI:10.1016/j.tim.2012.02.004
[31]
HONSA E S,COOPER V S,MHAISSEN M N,et al.RelA mutant enterococcus faecium with multiantibiotic tolerance arising in an immunocompromised host[J].MBIO,2017,8(1):e02116-e02124.
[32]
SHI C,LIU J Y,LI W J,et al.Expression of fluconazole resistance-associated genes in biofilm from 23 clinical isolates of Candida albicans[J].Brazilian Journal of Microbiology,2019,50(1):157-163.DOI:10.1007/s42770-018-0009-2
[33]
胡玢婕,赵付菊,赵虎.幽门螺杆菌生物膜形成与其耐药机制的相关性[J].检验医学,2014,29(8):865-870.
HU B J,ZHAO F J,ZHAO H.Study on the relationship between biofilm formation and drug resistance of helicobacter pylori[J].Laboratory Medicine,2014,29(8):865-870(in Chinese).DOI:10.3969/j.issn.1673-8640.2014.25
[34]
BOWLER L L,ZHANEL G G,BALL T B,et al.Mature pseudomonas aeruginosa biofilms prevail compared to young biofilms in the presence of ceftazidime[J].Antimicrobial Agents and Chemotherapy,2012,56(9):4976-4979.DOI:10.1128/AAC.00650-12
[35]
KHARA J S,OBUOBI S,WANG Y,et al.Disruption of drug-resistant biofilms using de novo designed shortα-helical antimicrobial peptides with idealized facial amphiphilicity[J].Acta Biomaterialia,2017,57:103-114.DOI:10.1016/j.actbio.2017.04.032
[36]
SEGEV-ZARKO L,SAAR-DOVER R,BRUMFELD V,et al.Mechanisms of biofilm inhibition and degradation by antimicrobial peptides[J].The Biochemical Journal,2015,468(2):259-270.DOI:10.1042/BJ20141251
[37]
ZHANG J Y,CHEN C X,CHEN J X,et al.Dual mode of anti-biofilm action of G3 against Streptococcus mutans[J].ACS Applied Materials&Interfaces,2020,12(25):27866-27875.
[38]
DE LA FUENTE-NUNEZ C,REFFUVEILLE F,MANSOUR S C,et al.D-enantiomeric peptides that eradicate wild-type and multidrug-resistant biofilms and protect against lethal Pseudomonas aeruginosa infections[J].Chemistry&Biology,2015,22(2):196-205.
[39]
XU D F,ZHANG Y,CHENG P,et al.Inhibitory effect of a novel chicken-derived anti-biofilm peptide on P.aeruginosa biofilms and virulence factors[J].Microbial Pathogenesis,2020,149:104514.DOI:10.1016/j.micpath.2020.104514
[40]
MOAZZEZY N,ASADI KARAM M R,RAFATI S,et al.Inhibition and eradication activity of truncatedα-defensin analogs against multidrug resistant uropathogenic Escherichia coli biofilm[J].PLoS One,2020,15(7):e0235892.DOI:10.1371/journal.pone.0235892
[41]
史鹏伟,高艳彬,卢志阳,等.抗菌肽LL-37对鲍曼不动杆菌生物膜的抑制作用[J].南方医科大学学报,2014,34(3):426-429.
SHI P W,GAO Y B,LU Z Y,et al.Effect of antibacterial peptide LL-37 on the integrity of Acinetobacter baumannii biofilm[J].Journal of Southern Medical University,2014,34(3):426-429(in Chinese).
[42]
DE BRUCKER K,DELATTIN N,ROBIJNS S,et al.Derivatives of the mouse cathelicidin-related antimicrobial peptide(CRAMP)inhibit fungal and bacterial biofilm formation[J].Antimicrobial Agents and Chemotherapy,2014,58(9):5395-5404.DOI:10.1128/AAC.03045-14
[43]
DI SOMMA A,MORETTA A,CANōC,et al.Antimicrobial and antibiofilm peptides[J].Biomolecules,2020,10(4):652.DOI:10.3390/biom10040652
[44]
SHANG D J,LIU Y,JIANG F Q,et al.Synergistic antibacterial activity of designed Trp-containing antibacterial peptides in combination with antibiotics against multidrug-resistant Staphylococcus epidermidis[J].Frontiers in Microbiology,2019,10:2719.DOI:10.3389/fmicb.2019.02719
[45]
PLETZER D,MANSOUR S C,HANCOCK R E W.Synergy between conventional antibiotics and anti-biofilm peptides in a murine,sub-cutaneous abscess model caused by recalcitrant ESKAPE pathogens[J].Plos Pathogens,2018,14(6):e1007084.DOI:10.1371/journal.ppat.1007084
[46]
ZHANG Y,HE X,CHENG P,et al.Effects of a novel anti-biofilm peptide CRAMP combined with antibiotics on the formation of Pseudomonas aeruginosa biofilms[J].Microbial Pathogenesis,2021,152:104660.DOI:10.1016/j.micpath.2020.104660
[47]
DOSLER S,KARAASLAN E.Inhibition and destruction of Pseudomonas aeruginosa biofilms by antibiotics and antimicrobial peptides[J].Peptides,2014,62:32-37.DOI:10.1016/j.peptides.2014.09.021
[48]
CHERNYSH S,GORDYA N,TULIN D,et al.Biofilm infections between Scylla and Charybdis:interplay of host antimicrobial peptides and antibiotics[J].Infection and Drug Resistance,2018,11:501-514.DOI:10.2147/IDR.S157847
[49]
HAKIMI ALNI R,TAVASOLI F,BARATI A,et al.Synergistic activity of melittin with mupirocin:a study against methicillin-resistant S.Aureus(MRSA)and methicillin-susceptible S.Aureus(MSSA)isolates[J].Saudi Journal of Biological Sciences,2020,27(10):2580-2585.DOI:10.1016/j.sjbs.2020.05.027
[50]
MUKHOPADHYAY S,BHARATH PRASAD A S,MEHTA C H,et al.Antimicrobial peptide polymers:no escape to ESKAPE pathogens-a review[J].World Journal of Microbiology&Biotechnology,2020,36(9):131.DOI:10.1007/s11274-020-02907-1
[51]
PALANISAMY N K,FERINA N,AMIRULHUSNI A N,et al.Antibiofilm properties of chemically synthesized silver nanoparticles found against Pseudomonas aeruginosa[J].Journal of Nanobiotechnology,2014,12(1):2.DOI:10.1186/1477-3155-12-2
[52]
FRANCI G,FALANGA A,GALDIERO S,et al.Silver nanoparticles as potential antibacterial agents[J].Molecules(Basel,Switzerland),2015,20(5):8856-8874.DOI:10.3390/molecules20058856
[53]
GAO J Y,NA H Y,ZHONG R,et al.One step synthesis of antimicrobial peptide protected silver nanoparticles:the core-shell mutual enhancement of antibacterial activity[J].Colloids and Surfaces B:Biointerfaces,2020,186:110704.DOI:10.1016/j.colsurfb.2019.110704
[54]
MENG Y S,HOU X C,LEI J X,et al.Multi-functional liposomes enhancing target and antibacterial immunity for antimicrobial and anti-biofilm against methicillin-resistant Staphylococcus aureus[J].Pharmaceutical Research,2016,33(3):763-775.DOI:10.1007/s11095-015-1825-9
[55]
BECKLOFF N,LAUBE D,CASTRO T,et al.Activity of an antimicrobial peptide mimetic against planktonic and biofilm cultures of oral pathogens[J].Antimicrobial Agents and Chemotherapy,2007,51(11):4125-4132.DOI:10.1128/AAC.00208-07
[56]
王家俊.非完美两亲性α螺旋肽的构效关系及其抗酶解活性的研究[D].博士学位论文.哈尔滨:东北农业大学,2019.
WANG J J.Studies on structure-function relationship and protease-resistant activity of imperfectly amphiphilicα-helical peptides[D].Ph.D.Thesis.Harbin:Northeast Agricultural University,2019.(in Chinese)
[57]
OKUDA K I,ZENDO T,SUGIMOTO S,et al.Effects of bacteriocins on methicillin-resistant Staphylococcus aureusbio film[J].Antimicrobial Agents and Chemotherapy,2013,57(11):5572-5579.DOI:10.1128/AAC.00888-13
[58]
ANSARI J M,ABRAHAM N M,MASSARO J,et al.Anti-biofilm activity of a self-aggregating peptide against Streptococcus mutans[J].Frontiers in Microbiology,2017,8:488.
[59]
BRANCATISANO F L,MAISETTA G,DI LUCA M,et al.Inhibitory effect of the human liver-derived antimicrobial peptide hepcidin 20 on biofilms of polysaccharide intercellular adhesin(PIA)-positive and PIA-negative strains of Staphylococcus epidermidis[J].Biofouling,2014,30(4):435-446.DOI:10.1080/08927014.2014.888062
[60]
PLETZER D,WOLFMEIER H,BAINS M,et al.Synthetic peptides to target stringent response-controlled virulence in a Pseudomonas aeruginosa murine cutaneous infection model[J].Frontiers in Microbiology,2017,1867.
[61]
FOLETTI C,KRAMER R A,MAUSER H,et al.Functionalized proline-rich peptides bind the bacterial second messenger c-di-GMP[J].Angewandte Chemie International Edition,2018,57(26):7729-7733.DOI:10.1002/anie.201801845
[62]
OVERHAGE J,CAMPISANO A,BAINS M,et al.Human host defense peptide LL-37 prevents bacterial biofilm formation[J].Infection and Immunity,2008,76(9):4176-4182.DOI:10.1128/IAI.00318-08
[63]
JACOBSEN A S,JENSSEN H.Human cathelicidin LL-37 prevents bacterial biofilm formation[J].Future Medicinal Chemistry,2012,4(12):1587-1599.DOI:10.4155/fmc.12.97
[64]
ARCIOLA C R,CAMPOCCIA D,BALDASSARRI L,et al.Detection of biofilm formation in Staphylococcus epidermidis from implant infections.Comparison of a PCR-method that recognizes the presence of ICA genes with two classic phenotypic methods[J].Journal of Biomedical Materials Research Part A,2006,76(2):425-430.
[65]
JENG W Y,KO T P,LIU C I,et al.Crystal structure of IcaR,a repressor of the TetR family implicated in biofilm formation in Staphylococcus epidermidis[J].Nucleic Acids Research,2008,36(5):1567-1577.DOI:10.1093/nar/gkm1176
[66]
ZHU C,TAN H L,CHENG T,et al.Humanβ-defensin 3 inhibits antibiotic-resistant Staphylococcus biofilm formation[J].The Journal of Surgical Research,2013,183(1):204-213.DOI:10.1016/j.jss.2012.11.048